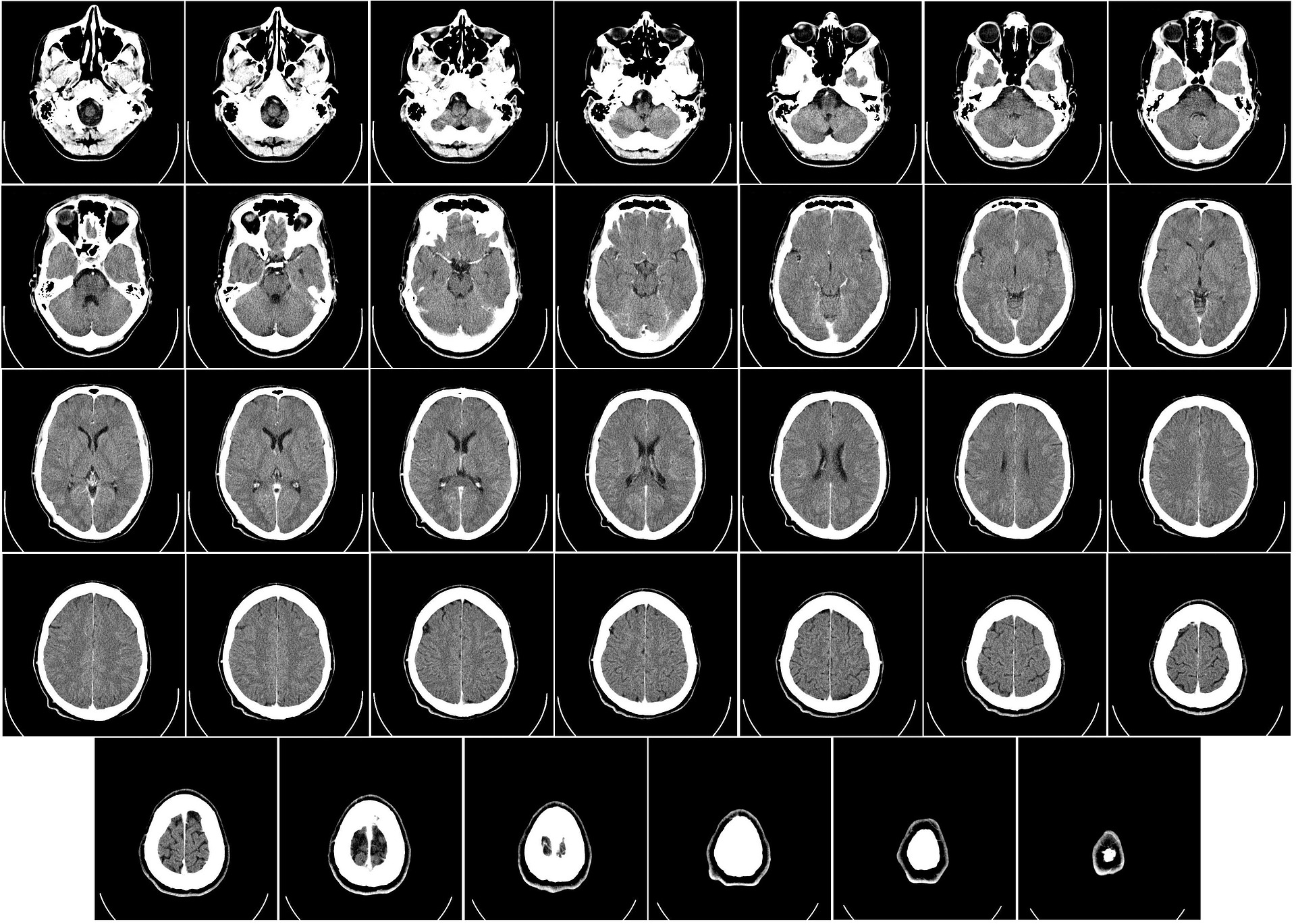
Wollen Mediziner und Biowissenschaftler einen detaillierten Blick in unser Inneres werfen, können sie sich unterschiedlicher bildgebender Verfahren bedienen. So können sie zum Beispiel herausfinden, wie groß unsere Leber ist, ob sie sich durch die Einnahme von Medikamenten verändert, ob die Niere entzündet ist oder ob sich im Gehirn ein Tumor befindet und dieser bereits Metastasen gebildet hat. Allerdings sind diese Analysen sehr zeitaufwändig, da eine Menge an Daten gesichtet und interpretiert werden muss.
„Die Auswertung von dreidimensionalen bildgebenden Verfahren ist sehr aufwändig“, erklärt Oliver Schoppe von der TU München. Er hat gemeinsam mit einem interdisziplinären Forschungsteam selbstlernende Algorithmen entwickelt, die künftig bei der Analyse biowissenschaftlicher Bilddaten helfen und die Auswertung entscheidend erleichtern können. Diese künstlichen neuronalen Netzwerke brauchen nur Sekunden, um Ganzkörperscans von Mäusen auszuwerten. Sie stellen die Organe, statt in verschiedenen Grau-Schattierungen in Farbe dar und können sie außerdem segmentieren.
Kernstück der AIMOS-Software (AI-based Mouse Organ Segmentation) sind künstliche neuronale Netze, die lernfähig sind. Ebenso wie das menschliche Gehirn. „Früher musste man Computerprogrammen genau sagen, was sie tun sollen“, erläutert Schoppe. „Neuronale Netze brauchen solche Vorgaben nicht mehr: Es genügt sie zu trainieren, indem man mehrmals eine Problemstellung und eine Lösung präsentiert. Die Algorithmen erkennen nach und nach die Muster und finden dann selbst die richtigen Lösungen.“
Selbstlernende Algorithmen trainieren
Trainiert wurden die Algorithmen im AIMOS-Projekt mit Hilfe von Mäusebildern. Sie sollten lernen, die Bildpunkte aus 3D-Ganzkörperscans bestimmten Organen zuzuordnen, beispielsweise Magen, Niere, Leber, Milz oder Gehirn. Aufgrund dieser Zuordnung kann das Programm dann die genaue Lage und Form darstellen.
„Glücklicherweise hatten wir Zugriff auf mehrere hundert Bilddatensätzen von Mäusen aus einem anderen Forschungsprojekt, die alle bereits von zwei Biologen interpretiert worden waren“, erinnert sich Schoppe. Zusätzlich nutzen die Forscher fluoreszenzmikroskopische 3D-Scans vom Institut für Tissue Engineering and Regenerative Medicine am Helmholtz Zentrum München.
Dort war es den Wissenschaftlern mit einer speziellen Technik bereits gelungen, verstorbene Mäuse vollständig zu entfärben. „Die transparenten Körper konnten Punkt für Punkt und Schicht für Schicht mit einem Mikroskop abgerastert werden.“ Die Abstände zwischen den Messpunkten hätten dabei nur sechs Mikrometer betragen – der Größe einer Zelle. Auch in diesen Datensätzen hätten Biologen die Organe lokalisiert.
Höhere Treffsicherheit durch Künstliche Intelligenz
Die neuen Algorithmen hätten schneller gelernt als erwartet, berichtete Schoppe nach einer Präsentation am TranslaTUM. „Wir haben nur etwa zehn Ganzkörperscans gebraucht, dann konnte die Software die Analyse der Bilddaten allein bewerkstelligen – und zwar innerhalb von Sekunden. Ein Mensch braucht dafür Stunden.“
Die Forscher überprüften die Zuverlässigkeit der Künstlichen Intelligenz anschließend mit Hilfe von 200 weiteren Ganzkörper-Scans von Mäusen. „Das Ergebnis zeigt, dass selbstlernende Algorithmen bei der Auswertung biologischer Bilddaten nicht nur schneller, sondern auch treffsicherer sind als der Mensch“, resümiert Prof. Bjoern Menze, Leiter der Image-Based Biomedical Modeling Group am TranslaTUM der Technischen Universität München TUM.
„Viel Zeit sparen”
In Zukunft soll die Intelligente Software vor allem in der Grundlagenforschung eingesetzt werden. „Bildaufnahmen von Mäusen werden dringend benötigt, zum Beispiel um die Wirkweise von neuen Medikamenten zu untersuchen, bevor sie beim Menschen zum Einsatz kommen. Die Auswertung von Bilddaten mit selbstlernenden Algorithmen kann künftig viel Zeit sparen“, betont Menze.
Unterstützung bekamen sie Forscher bei ihrem Projekt durch das Bundesministerium für Bildung und Forschung (BMBF) im Rahmen der Software Campus Initiative, die Deutsche Forschungsgemeinschaft (DFG) über den Exzellenzcluster Munich Cluster for Systems Neurology (SyNergy) sowie ein Forschungsstipendium und durch das TUM Institute for Advanced Study aus Mitteln der Exzellenzinitiative und der Europäischen Union. Außerdem wurde die Arbeit, die im Fachmagazin Nature veröffentlicht wurde, durch die Fritz Thyssen Stiftung gefördert und NVIDIA unterstützte die Arbeiten über das GPU Grant Programm.