Seit Ausbruch des Corona-Virus wird rund um die Welt nach Heilmitteln und Impfstoffen geforscht. Es vergeht kaum ein Tag, an dem nicht irgendein Labor Fortschritte vermeldet. Dennoch, verlässliche Medikamente gibt es bis jetzt noch nicht. Wissenschaftler des Max-Planck-Institut für Biophysik in Frankfurt am Main wollen nun das Oberflächenprotein des neuartigen Coronavirus analysieren. Dadurch hoffen sie, Bindungsstellen für Impfstoffe und Medikamente aufzuspüren.
Virus sieht aus wie eine Krone
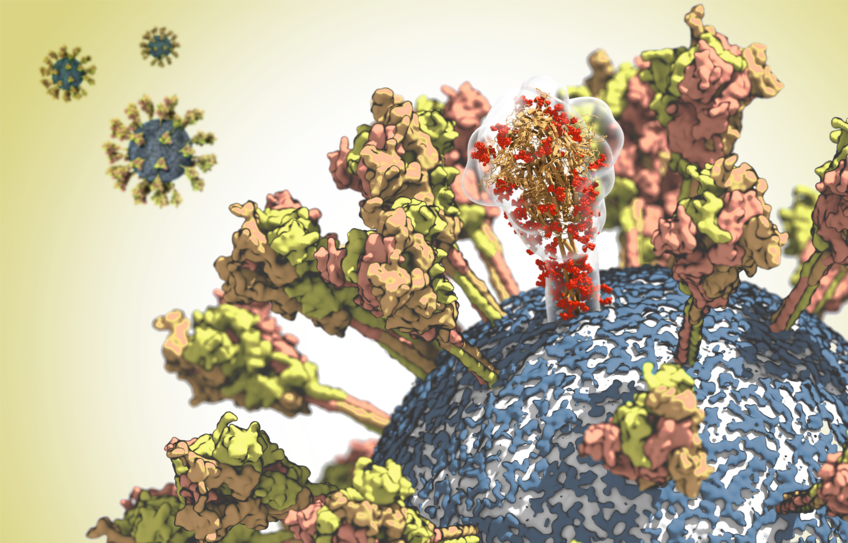
Das Coronavirus heißt wörtlich übersetzt Kronenvirus. Seinen Namen verdankt es dem Glykoprotein Spike. Dieses Molekül steht wie Zacken einer Krone von der Virushülle ab. Die Forscher wollen nun die Struktur dieses Proteins genauer untersuchen und so potenzielle Ziele für Antikörper und Hemmstoffe identifizieren. Genaue Informationen, wo und auf welche Weise das SARS-CoV-2-Virus angreifbar wäre, sind eine wichtige Voraussetzung für die Entwicklung neuer Impfstoffe und Medikamente.
Krone – Waffe und Achillesferse zugleich
Das Spike-Protein ist die Waffe, mit der das Coronavirus Zellen befällt. Es bindet vor allem an einen Rezeptor auf der Oberfläche menschlicher Zellen namens ACE2. Auf diese Weise gelingt es dem Virus mit der Zellmembran zu verschmelzen und sein Erbgut ins Zellinnere zu entlassen. Genau diese Spike-Protein könnte aber nicht nur die beste Waffe des Virus sein, sondern auch seine Achillesferse, glauben die Max-Planck-Wissenschaftler. So hoffen, somit seine Schwachstelle gefunden zu haben.
Aufgrund der „exponierten Lage“, das heißt indem es von der Virushülle absteht, stellt das Spike-Protein auch einen Angriffspunkt für das Immunsystem dar. So könnten Antikörper das Virus anhand des Spike-Proteins erkennen. Sich daran binden und es so als Ziel für Immunzellen markieren, erklären die Forscher. Allerdings habe das Virus einen Trick auf Lager: Mithilfe von Zuckermolekülen versuche es, seine Spike-Proteine vor den Immunzellen zu verbergen.
Zuckerhülle zur Verteidigung
Um diesen Zucker-Schutzschirm auszuschalten, analysieren die Wissenschaftler nicht nur das Spike-Protein selbst, sondern auch die Membranhülle des Virus und den Schutzschirm. Auf diese Weise wollen sie über die bislang existierenden statischen Strukturen hinaus berechnen. Sie erforschen, wie sich die Spike-Proteine auf der Virusoberfläche bewegen und ihre Form verändern – „und das mit einer Genauigkeit, die der Größe eines Atoms entspricht“.
Die Berechnungen dieses dynamischen Modells des Spike-Proteins seien „extrem aufwändig“ würden aber auch winzigste Details der Proteinstruktur enthüllen. „Wir brauchen dazu die Supercomputer der Max-Planck-Gesellschaft mit ihrer enormen Rechenleistung“, erklärt Gerhard Hummer, Direktor am Max-Planck-Institut für Biophysik.
Ziel des Modells ist es, Domänen aufzuspüren, an die Antikörper verlässlich binden können und auch, Bindungsstellen für Hemmstoffe zu entdecken. Diese sollen dann mit den Bindungseigenschaften bereits existierender Medikamente am Computer verglichen werden, um so Wirkstoffe zu identifizieren, die das Spike-Protein blockieren können. „Medikamente, die bereits auf dem Markt sind, umzuwidmen, geht natürlich viel schneller als neue Wirkstoffe ausfindig zu machen und in langwierigen klinischen Studien zu testen“, sagt Hummer.
Titelbild: Oberfläche des CoV-2-Virus. Ein Molekül des spike-Proteins ist durchscheinend dargestellt, um seine komplexe räumliche Struktur hervorzuheben. © MPI f. Biophysik