Während weltweit immer mehr Impfstoffe gegen das Coronavirus SARS-CoV-2 zum Einsatz kommen, scheint die Forschung nach Medikamenten gegen COVID-19 zu keinen entscheidenden Ergebnissen zu führen. Verschiedene, bereits existierende Wirkstoffe, auf die die Mediziner große Hoffnungen gesetzt hatten, haben sich in der Praxis nicht bewährt. So wurde ein HIV-Medikament mit der Wirkstoffkombination Lopinavir / Ritonavir als nicht wirksam abgehakt. Ebenso wie mehrere Malaria-Medikamente mit den Wirkstoffen Chloroquin und Hydroxychloroquin, die die Krankheit teilweise sogar verschlimmerten. Ein Forschungsteam am DESY hat nun mit Hilfe der Röntgenlichtquelle PETRA III aber offenbar Fortschritte gemacht und konnte mehrere Kandidaten für Wirkstoffe gegen das SARS-CoV-2 identifizieren.
Sie testeten in kurzer Zeit fast 6.000 Wirkstoffe, die bereits für die Behandlung anderer Krankheiten eingesetzt werden und konnten nach der Messung von rund 7000 Proben insgesamt 37 Stoffe identifizieren, die an die Hauptprotease (Mpro) des SARS-CoV-2-Virus binden. Dabei bremsten sieben Stoffe die Vermehrung des Virus und könnten eine weitere Verbreitung im Körper des Patienten stoppen. Zwei dieser Stoffe werden aktuell bereits in präklinischen Studien weiter untersucht. „Das vermutlich größte Wirkstoffscreening dieser Art brachte zudem eine neue Bindungsstelle an der Hauptprotease des Virus zu Tage, an der Medikamente ankoppeln können“, schreiben die Wissenschaftler im renommierten Fachmagazin „Science“ online.
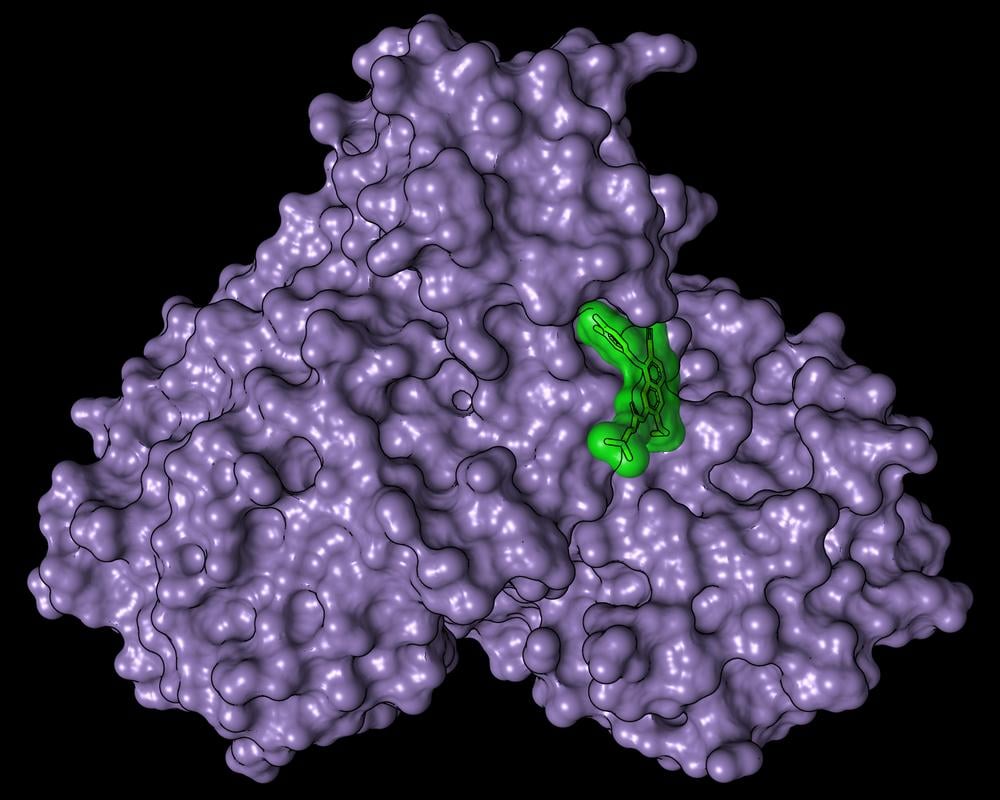
Da Viren sich nicht alleine vermehren können, schleusen sie ihr Erbgut in die Zellen ihres Wirts ein und bringen diese dazu, neue Viren herzustellen. Proteinen wie der Hauptprotease des Virus fällt hierbei eine entscheidende Rolle zu. Sie zerschneidet Proteinketten in kleinere Teile, die nach dem Bauplan des Viruserbguts von der Wirtszelle hergestellt wurden und für die Vermehrung des Virus notwendig sind. Wird diese Hauptprotease nun blockiert, kann sich das Virus nicht mehr vermehren, und die Infektion ist besiegt.
Dreidimensionale räumliche Struktur von Proteinen
Mit der Forschungslichtquelle PETRA III kann die dreidimensionale räumliche Struktur von Proteinen atomgenau dargestellt werden. Diese Fähigkeit nutzten die Forscher um DESY-Physiker Alke Meents und untersuchten mehrere tausend bekannte Wirkstoffe aus Bibliotheken des Fraunhofer-Instituts für Translationale Medizin und Pharmakologie und der italienischen Firma Dompé Farmaceutici SpA daraufhin, ob und wie sie an die Hauptprotease „andocken“ und dann auch blockiert werden könnten. Wie der Schlüssel in ein Schloss passe dabei das Wirkstoffmolekül in ein Bindungszentrum der Protease, erklären sie.
Der Vorteil der Wirkstoffbibliothek ist, dass geeignete Kandidaten zur Bekämpfung von SARS-CoV-2 erheblich schneller in klinischen Studien eingesetzt werden könnten, da alle Stoffe bereits für die Behandlung von Menschen zugelassen sind oder sich zumindest schon in verschiedenen Erprobungsphasen befinden. So könnte man Monate oder sogar Jahre der Wirkstoffentwicklung einsparen. „Mit Hilfe eines Hochdurchsatzverfahrens haben wir insgesamt 37 Wirkstoffe finden können, die eine Bindung mit der Hauptprotease eingehen“, sagt Meents, der die Experimente initiierte.
Als nächstes untersuchten Forscher am Bernhard-Nocht-Institut für Tropenmedizin, inwieweit die Wirkstoffe in Zellkulturen die Virusvermehrung hemmen oder gar verhindern, und wie verträglich sie für die Wirtszellen sind. Am Ende blieben sieben Wirkstoffe übrig, von denen sich zwei besonders hervortaten. „Die Wirkstoffe Calpeptin und Pelitinib zeigten die deutlich höchste Antiviralität bei guter Zellverträglichkeit“, erklärt DESY-Forscher Sebastian Günther, Erstautor der „Science“-Veröffentlichung. „Unsere Kooperationspartner haben daher bereits präklinische Untersuchungen mit diesen beiden Wirkstoffen begonnen.“
Bisher unbekannte Bindungsstelle gefunden
Bei ihrem Wirkstoffscreening erlebten die Wissenschaftler auch eine große Überraschung. Sie fanden eine Bindungsstelle an der Hauptprotease, die bis dahin noch völlig unbekannt war. „Es war nicht nur eine positive Überraschung, dass wir eine neue Bindestelle für Medikamente an der Hauptprotease entdecken konnten – ein Ergebnis, das man wirklich nur an einer Synchrotronlichtquelle wie PETRA III erzielen kann –, sondern dass sogar einer der beiden heißen Wirkstoffkandidaten genau an diese Stelle bindet“, sagt Christian Betzel vom Exzellenzcluster CUI der Universität Hamburg, Mitinitiator der Studie.
„Eine besondere Stärke unserer Methode des Röntgenscreenings im Vergleich zu anderen Screeningmethoden ist, dass wir als Ergebnis die dreidimensionale Struktur der Protein-Wirkstoff-Komplexe erhalten und damit die Bindung der Wirkstoffe an das Protein auf atomarer Ebene bestimmen können“, erläutert Patrick Reinke, DESY-Forscher und Koautor der Veröffentlichung. „Selbst wenn die beiden aussichtsreichsten Kandidaten es nicht in klinische Studien schaffen sollten, so bilden die 37 Stoffe, die an die Hauptprotease binden, eine wertvolle Datenbasis für darauf aufbauende Medikametenentwicklungen.“
An den Arbeiten sind neben DESY-Wissenschaftlerinnen und -Wissenschaftlern auch Forscherinnen und Forscher der Universitäten Hamburg und Lübeck, des Bernhard-Nocht-Instituts für Tropenmedizin, des Fraunhofer-Instituts für Translationale Medizin und Pharmakologie, des Heinrich-Pette-Instituts, des European XFEL, des Europäischen Laboratoriums für Molekularbiologie EMBL, der Max-Planck-Gesellschaft, des Helmholtz-Zentrums Berlin und weiteren Institutionen beteiligt. Zusätzlich zu den Experimenten an der Messstation P11 wurden auch Messungen an den EMBL-Messstationen P13 und P14 an PETRA III durchgeführt.
Titelbild: Oberflächendarstellung der Corona-Hauptprotease (violett) mit gebundenem Wirkstoff Pelitinib (grün). Bild: DESY, Sebastian Günther
Weitere Artikel zum Thema Coronapandemie und SARS-CoV-2 finden Sie hier.

