Das Coronavirus Sars-CoV-2 benötigt Spikeproteine auf seiner Oberfläche, um an spezifische Rezeptoren auf der Oberfläche menschlicher Zellen zu binden und die Zellen zu infizieren. Diese stachelförmigen Strukturen stehen im Mittelpunkt der Entwicklung von Impfstoffen, denn sie induzieren eine Immunantwort beim Menschen. Intensiv untersuchen Forschende das Virus und insbesondere dessen Oberflächenstruktur, um Erkenntnisse sowohl für die Impfstoffe als auch für die Entwicklung wirksamer Therapeutika zur Behandlung infizierter Patientinnen und Patienten zu gewinnen. Von großer Bedeutung ist hierbei die Kenntnis der räumlichen Strukturen, die für Prozesse wie beispielsweise das Eindringen in die Zielzellen bedeutsam sind.
Wissenschaftlerinnen und Wissenschaftler des Max-Planck-Instituts für Biophysik, Paul-Ehrlich-Instituts, des European Molecular Biology Laboratory (EMBL) und der Goethe Universität Frankfurt haben mit hochauflösenden bildgebenden und computergestützten Verfahren das Spikeprotein in seiner natürlichen Umgebung analysiert. Dabei haben sie überraschende Erkenntnisse gewonnen, darunter eine nicht vermutete Bewegungsfreiheit, so einen Pressebericht des Max-Planck Institut.
Mithilfe modernster Elektronenmikroskopie am EMBL wurden 266 Kryo-Tomogramme von etwa 1000 verschiedenen Viren erstellt, die jeweils durchschnittlich 40 Spikes auf ihrer Oberfläche trugen. Durch Subtomogramm-Mittelung und Bildverarbeitung konnten von insgesamt 40.000 Spikeswichtige Strukturinformationen gewonnen werden.
Flexibler Stiel
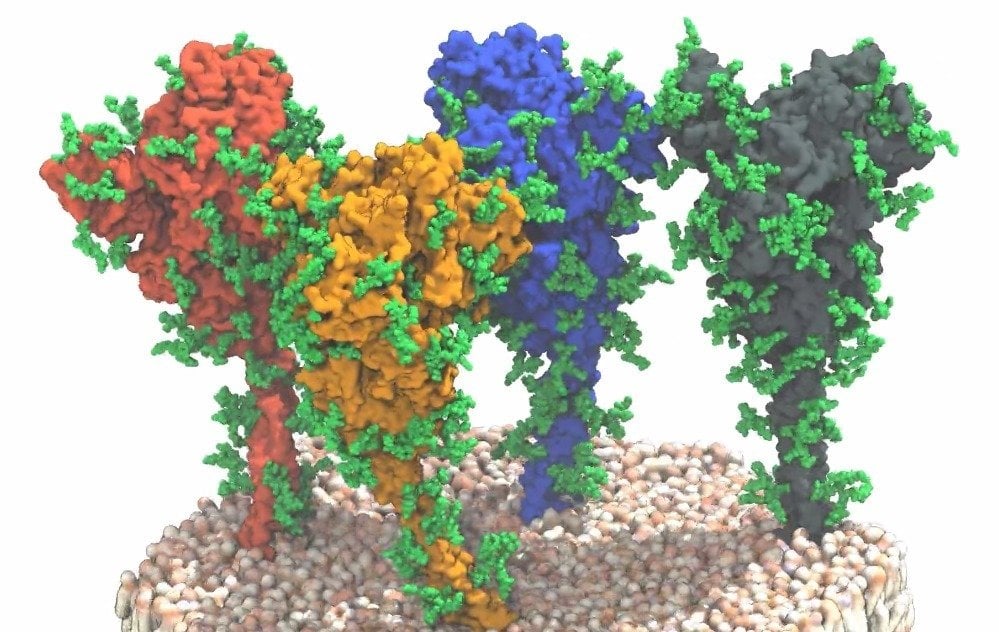
Ein für die Impfstoffentwicklung erfreuliches Ergebnis: “Der obere Kugel- bzw. V-förmige Teil des Spikes weist unter natürlichen Bedingungen eine Struktur auf, die von rekombinanten Proteinen, die für die Impfstoffentwicklung genutzt werden, gut wiedergegeben wird. Befunde über den Stiel, mit dem der globuläre Teil des Spikeproteins auf der Virusoberfläche fixiert ist, waren jedoch neu”, sagt Martin Beck, Gruppenleiter am EMBL und Direktor am Max-Planck-Institut für Biophysik. “Wir haben erwartet, dass der Stiel ziemlich starr ist. Aber unsere Modelle und auch die Bilder zeigen, dass er im Gegenteil äußerst flexibel ist”, fügt Gerhard Hummer vom Max-Planck-Institut für Biophysik und von der Universität Frankfurt hinzu. In den Aufnahmen stand er selten gerade auf der Membran, sondern war in alle Richtungen geneigt. Das Team identifizierte vier verschiedene Domänen im Stiel, die sie als „Hüft-“, „Knie“-, „Knöchel“-Domäne, und schließlich als in die Membran eingebettete „Fuß“-Domäne bezeichneten. Durch Kombination von Molekulardynamik-Simulationen und Kryo-Tomografie haben sie nachgewiesen, dass diese Abschnitte Biegebewegungen ausführen können.
Die Daten zeigen, dass der globuläre Anteil des Spikeproteins, der den Rezeptorbindungsbereich und die für die Fusion mit der Zielzelle erforderlichen Bestandteile enthält, mit einem flexiblen Stiel verbunden ist. „Wie ein Ballon an einer Schnur scheinen sich die Spikes auf der Oberfläche des Virus zu bewegen und so den Rezeptor für das Andocken an der Zielzelle suchen zu können“, beschreibt Jacomine Krijnse Locker vom Paul-Ehrlich-Institut die Ergebnisse. Die Analysen zeigten schließlich, dass der Stiel mit vielen Glykanketten ausgestattet ist. Diese könnte dem Stiel zudem eine Art schützenden Mantel aus Kohlenhydraten verleihen und ihn so vor neutralisierenden Antikörpern verstecken – eine weitere wichtige Erkenntnis auf dem Weg zu wirksamen Impfstoffen und Medikamenten.
Titelbild: Vier Spike-Proteine auf der Membranoberfläche von Sars-CoV-2. © MPI f. Biophysik/ von Bülow, Sikora, Hummer